Inventio
Vol. 20, núm. 50, 2024
doi: http://doi.org/inventio/10.30973/2024.20.50/1
Identificación de péptidos lineales como prospectos inmunogénicos in silico contra Anaplasma marginale
Identification of linear peptides as in silico immunogenic leads against Anaplasma marginale
Cristopher Favian Guadarrama Albarrán
orcid: 0009-0001-3575-6014, favian-19999@outlook.es
Maestría en Ciencias en Biotecnología, Universidad Politécnica del Estado de Morelos (upemor)
Elizabeth Salinas Estrella
orcid: 0000-0002-0503-6577, salinas.elizabeth@inifap.gob.mx
Centro Nacional de Investigación Disciplinaria en Salud Animal e Inocuidad (cenid-sai), Instituto Nacional de Investigaciones Forestales, Agrícolas y Pecuarias (inifap)
Mayra Elizeth Cobaxin Cárdenas
orcid: 0000-0001-9984-2715, cobaxin.mayra@inifap.gob.mx
Centro Nacional de Investigación Disciplinaria en Salud Animal e Inocuidad (cenid-sai), Instituto Nacional de Investigaciones Forestales, Agrícolas y Pecuarias (inifap)
Sergio Darío Rodríguez Camarillo
orcid: 0000-0003-1316-5739, rodriguez.sergio@inifap.gob.mx
Centro Nacional de Investigación Disciplinaria en Salud Animal e Inocuidad (cenid-sai), Instituto Nacional de Investigaciones Forestales, Agrícolas y Pecuarias (inifap)
resumen
Anaplasma marginale es un patógeno con una amplia diversidad genética, la cual ha dificultado el desarrollo de vacunas eficientes para la prevención y control de la anaplasmosis bovina. Mediante el uso de programas bioinformáticos se realizó una predicción de péptidos lineales de proteínas de membrana que contuvieran en su secuencia epítopos b, para su posible proposición como candidatos a inmunógenos. Este trabajo es una aproximación in silico para la posterior síntesis en formato péptidos multiantigénicos sintéticos (map) y su subsecuente evaluación in vivo, de forma que se analice la efectividad en la generación de una respuesta inmune protectora frente a un desafío heterólogo con cepas de Anaplasma marginale.
palabras clave
anaplasmosis bovina, diagnóstico, inmunobioinformática, inmunoprotección, vacunología
abstract
Anaplasma marginale is a pathogen with a wide genetic diversity, which has hindered the development of efficient vaccines for the prevention and control of bovine anaplasmosis. Using bioinformatics programs, a prediction of linear peptides of membrane proteins containing b epitopes in their sequence was carried out for their possible proposal as immunogen candidates. This work is an in silico approach for the subsequent synthesis in synthetic multiantigenic peptide (map) format and its subsequent in vivo evaluation, in order to analyze the effectiveness in the generation of a protective immune response against a heterologous challenge with strains of Anaplasma marginale.
key words
bovine anaplasmosis, diagnosis, immunobioinformatics, immunoprotection, vaccinology
Recepción: 14/06/23. Aceptación: 19/01/24. Publicación: 05/07/24.
Introducción
La anaplasmosis bovina es una enfermedad causada por Anaplasma marginale, organismo patógeno de bovinos, el cual ha sido el único de la familia Anaplasmataceae reportado en México. Ésta es una bacteria de tipo Gram negativa, del género de las rickettsias y del genogrupo ii de las Ehrlichias. Carece de citoplasma y retículo endoplasmático, tiene una forma cocoide de aproximadamente 0.3 μm de diámetro y es intraeritocítica obligada (Rodríguez Carrillo et al., 2003).
En México, se ha determinado, por medio de serología y pruebas moleculares, una prevalencia del 50% en las regiones tropicales y subtropicales (Rodríguez et al., 2009). Las cepas mexicanas de A.< marginale muestran una amplia diversidad genética de la proteína msp1a, la cual cumple la función de adhesina de los cuerpos iniciales hacia los eritrocitos y las células epiteliales del intestino de la garrapata. Esta diversidad revela el importante papel del transporte de ganado en la transmisión, distribución y mantenimiento de la enfermedad entre los hatos bovinos del país, y refuerza la propuesta de vacunas regionales para controlar los brotes de anaplasmosis (Salinas-Estrella et al., 2022). Asimismo, hace patente la necesidad de desarrollar nuevas vacunas utilizando otras proteínas presentes en la membrana de A. marginale que tengan un alto grado de conservación.
Por otro lado, esta diversidad es el principal obstáculo para el desarrollo de una vacuna efectiva, cuyo objetivo es que los bovinos desarrollen una respuesta inmune protectora tipo th1. La respuesta th1 se caracteriza por la presencia de altos títulos de igg2, debido a que las células th1 son las encargadas de la producción de la citocina il-2 e interferón gamma, que a su vez estimulan la síntesis del isotipo igg2 e interferón (ifn-γ) por las células b y los macrófagos, los cuales son capaces de fagocitar a los eritrocitos infectados por A. marginale. A diferencia del isotipo igg2, el isotipo igg1 no es capaz de mediar la fagocitosis por neutrófilos o monocitos (Barigye et al., 2004).
Es por ello que, idealmente, las vacunas que se propongan en adelante mediante la utilización de estrategias bioinformáticas deben asegurar que la respuesta inmune que desencadenen sea una respuesta protectora, con la cual el sistema inmune de los bovinos deberá reconocer epítopos conservados en las proteínas de las diferentes cepas circulantes en México. De esta forma, deben asegurar que las características de virulencia propias de A. marginale —como los picos de rickettsemia originados por el cambio en la expresión cíclica de la proteína msp2 o la enfermedad ocasionada por la infección de múltiples cepas— no sean un impedimento para que las células del sistema inmune monten una respuesta adaptativa fuerte y prolongada a través del tiempo, con la cual podrán evitar la presentación del cuadro clínico ocasionado por la replicación de A. marginale en los eritrocitos bovinos.
Objetivo
Identificar péptidos inmunogénicos en A. marginale a través de herramientas bioinformáticas para la proposición de un posible candidato a inmunógeno contra la anaplasmosis bovina.
Materiales y métodos
Se realizó una búsqueda del genoma de las cepas de A. marginale registradas en el Centro Nacional de Información Biotecnológica (ncbi) en Estados Unidos. Entre las veinticuatro cepas resultantes (National Library of Medicine, sf) se identificaron las siete cepas mexicanas, con números de acceso vtww01000000, vtso01000000, vtcz01000000, qliv01000000, vtcx01000000, vtwv01000000 y vtcy01000000, así como la de St. Maries, con número de acceso nc_004842, cuyo genoma ya ha sido secuenciado en su totalidad.
Se localizaron todas las proteínas presentes en la membrana de A. marginale de la cepa St. Maries y se procedió a realizar una búsqueda específica de las siguientes proteínas de membrana: bam, sec, mur, tat, omp, vir, msp, rod, glm y ple. Se descargaron las tablas de todas las proteínas encontradas y, mediante el uso de MobaxTerm (simulador de Linux), se filtraron los nombres de las proteínas que se repetían en la cepa St. Maries. Posteriormente, se seleccionó una proteína multispecies que estuviera presente en todas las cepas mexicanas.
Para visualizar el grado de conservación de la proteína seleccionada entre las cepas mexicanas y la de St. Maries se realizó un alineamiento múltiple con Clustal Omega. Por otra parte, se hizo el análisis de topología de las proteínas, así como de hidrofobicidad, antigenicidad y dominios proteicos mediante los programas bioinformáticos tmhmm, protter, Phobius, Phyre2 y smart-pfam. De esta manera se pudo predecir si la proteína era expuesta, transmembranal o intracelular.
Finalmente, se utilizaron los programas bioinformáticos iedb Analysis Resource, BepiPred, abcpred y svmtrip para la predicción de epítopos b. Estos programas cuentan con un umbral de predicción de cero a uno y, en el caso de iedb, se muestra un puntaje (score) máximo y mínimo de la proteína, ya establecido por el método de predicción BepiPred Linear Epitope Prediction, por lo que se tomaron en cuenta sólo aquellos epítopos b lineales que presentaron un valor entre 0.5 y uno, con una longitud de diez a dieciséis aminoácidos, para la definición y selección de los péptidos que serían propuestos como candidatos a inmunógenos (figura 1).
Figura 1
Proceso de identificación de péptidos lineales como prospectos inmunogénicos in silico contra Anaplasma marginale
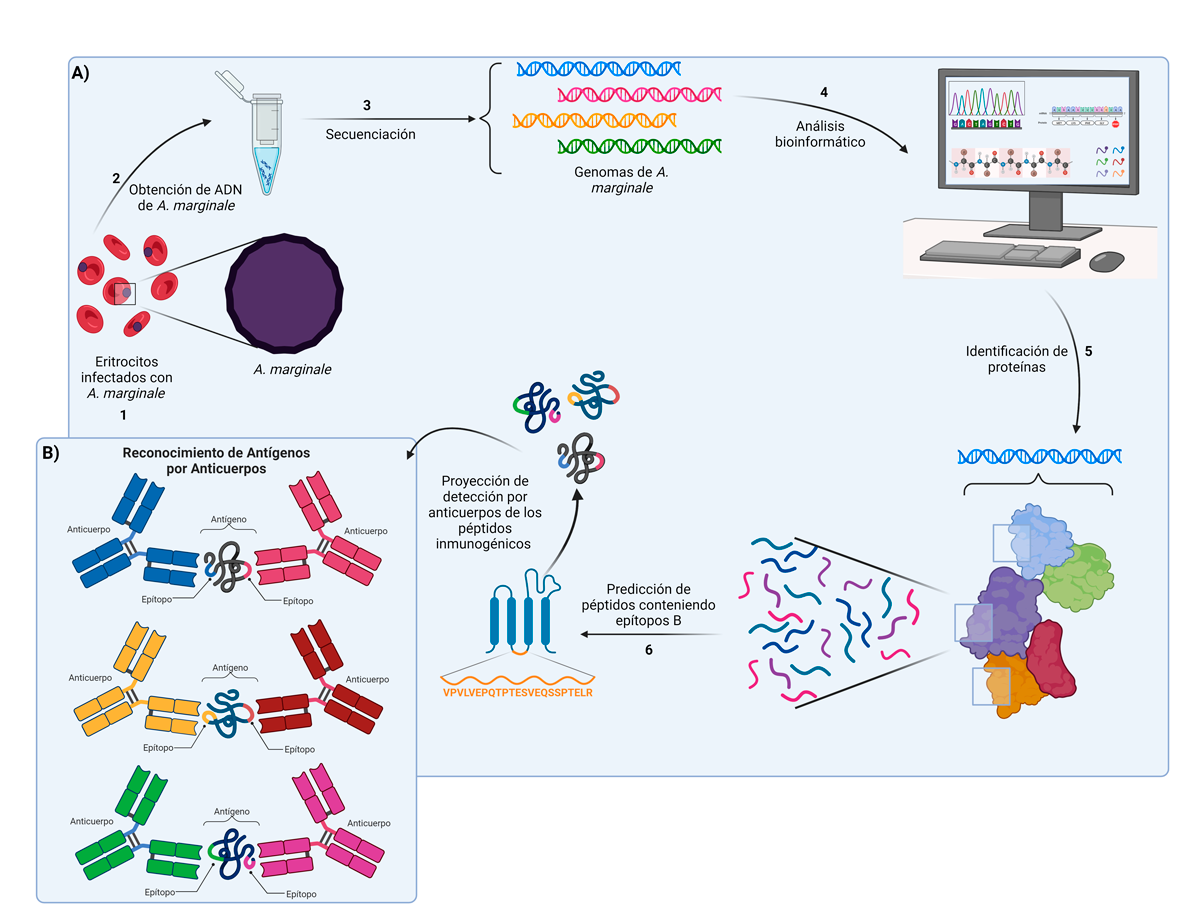
A) A partir de eritrocitos de bovinos infectados con A. marginale (1) se obtiene el material biológico (adn) del patógeno (2) para secuenciarse (3). Por medio de herramientas bioinformáticas, se realiza el análisis de las secuencias de las diferentes cepas registradas (4) para, de esta forma, encontrar dominios de proteínas (5) importantes para las funciones prioritarias de A. marginale (adhesión, respiración, transporte de nutrientes, entre otras). A partir de las secuencias de proteínas, se utilizan diferentes algoritmos que ayudan a predecir porciones cortas (péptidos) que contengan epítopos b (6), con base en la expresión en membrana, hidrofobicidad y antigenicidad de las proteínas y, en específico, de los péptidos predichos.
B) La finalidad es seleccionar péptidos lineales con el mayor potencial de ser antigénicos y reconocidos por
anticuerpos en su conformación final en las proteínas nativas. Todos estos análisis son realizados in silico, es decir, hechos por computadora o por vía de simulación computacional.
Fuente: elaboración propia.
Resultados y discusión
Se escogieron los epítopos b con mayor puntaje en los distintos programas bioinformáticos, cada uno con un distinto umbral de predicción. Los epítopos con diez a dieciséis aminoácidos y con el mayor puntaje considerado por cada programa fueron seleccionados para los análisis posteriores, con la finalidad de aumentar la probabilidad de detección por los anticuerpos bovinos, debido al tamaño del sitio de unión al antígeno de la región variable que posee el isotipo igg de tipo monomérico.
Se consideraron los epítopos egyidsypssksidya (posición 113-129) y aptirepilggngeis (posición 282-298) de las proteínas bamd y secd de dieciséis aminoácidos predichos por el programa abcpred, debido a que se traslapaban con los predichos con el programa iedb (tabla 1). Por otra parte, el epítopo gfenrdasvgvpi (posición 133-146) de la proteína tatc de trece aminoácidos, predichos por el programa BepiPred, se tomó en cuenta debido a que se encontraba traslapándose con el predicho con iedb al igual que los anteriores (tabla 1). Todos los epítopos se encuentran al exterior de la membrana, de acuerdo con los resultados obtenidos en los programas VaxiJen, Protscale, tmhmm, Phobius, protter, Phyre2, Interpro y smart-pfam.
Tabla 1
Selección de los epítopos b predichos mediante programas bioinformáticos
| Cepas | Proteínas transmembranales analizadas | Predicción de epítopos b y programa bioinformático utilizado | Propuesta de epítopo b como candidato vacunal contra anaplasmosis bovina |
| St. Maries mex-14-010-01 mex-30-193-01 mex-01-001-01 mex-17-017-01 mex-31-096-01 mex-30-184-02 mex-15-099-01 | |||
| tatc | iedb nrdasvg abcpred vglissktlagkrrva BepiPred gfenrdasvgvpi svmtrip vaivaiftlaailt |
gfenrdasvgvpi | |
| secd | iedb pilggngeis abcpred aptirepilggngeis BepiPred repilggngeisgrfsmseak svmtrip llevdfheylkgrl |
repilggngeisgrfsmseak | |
| bamd | iedb gyidsypssksi abcpred egyidsypssksidya BepiPred pvaaasfaeegvh svmtrip rhlgeiskrlaavq |
egyidsypssksidya |
Fuente: elaboración propia.
Con base en los análisis bioinformáticos sobre las proteínas bamd, secd y tatc, los péptidos derivados de éstas pueden ser considerados como candidatos a inmunógenos. También se determinó la distribución de la proteína en la membrana y, mediante el análisis de la secuencia con programas bioinformáticos, se determinó que los epítopos de secuencia egyidsypssksidya, aptirepilggngeis y gfenrdasvgvpi, predichos con abcpred, BepiPred, y los traslapados con los de iedb, gyidsypssksi, pilggngeis y nrdasvg, se encuentran expuestos, por lo cual se puede utilizar la secuencia traslapada de éstos como péptidos inmunogénicos, por su potencial para ser reconocidos por anticuerpos e, idealmente, generar una respuesta inmune protectora th1.
Se encontraron similitudes con la metodología usada en este trabajo en el estudio publicado por Ramírez Rueda (2007), quien realizó una predicción de secuencias con actividad inmunogénica, útiles para el diseño de un modelo de posible vacuna contra la malaria, en el cual utilizaba los programas tappred, Propred i, Propred, ctlpred y abcpred, para la predicción de epítopos, y tmhmm para la predicción de la posición del epítopo, es decir, si éste se encontraba expuesto o estaba internalizado.
Como se puede apreciar en los resultados obtenidos en la predicción de epítopos b como posibles candidatos vacunales, se emplearon cuatro programas disponibles en internet, de los cuales sólo abcpred coincide como la herramienta utilizada en el presente trabajo. Además, en la predicción de la posición del epítopo se utilizaron más programas bioinformáticos, lo cual genera una mayor probabilidad de que el péptido efectivamente esté expuesto, y también se utilizaron programas para predecir los dominios proteicos, de tal manera que si el epítopo está dentro del dominio, es más probable que pueda ser inmunogénico.
A pesar de los interesantes resultados obtenidos, la cobertura de este trabajo es meramente de análisis y determinación in silico, con base en las propiedades antes descritas de los programas usados y las características buscadas de las diferentes proteínas conservadas entre cepas de A. marginale. La comprobación de la efectividad de los epítopos encontrados como estimuladores de una respuesta inmune protectora contra A. marginale requiere, en primer lugar, de la síntesis de dichos epítopos en forma de péptidos sintéticos multiantigénicos (map, por sus siglas en inglés), para ser inoculados en bovinos junto con un adyuvante, con la intención de que esos péptidos sean reconocidos por las células del sistema inmune, cuyos productos o moléculas pudieran identificar eficazmente los mismos sitios de unión en proteínas nativas de A. marginale.
La respuesta inmune protectora no se podría dar por establecida hasta que los bovinos previamente inoculados fueran desafiados con una cepa viva de A. marginale y se comprobara que han sobrevivido a la infección sin desarrollar signos de enfermedad y sin presentar ciclos de rickettsemia en los meses posteriores. Para ello, el estado de salud de los bovinos inoculados se debería evaluar mediante un seguimiento clínico cercano. Éste debe incluir: a) la evaluación de la temperatura rectal (cuyo aumento indica un proceso infectivo en desarrollo detectado por el sistema inmune); b) la observación al microscopio de frotis sanguíneo, para determinar la presencia del patógeno y para la cuantificación del porcentaje de eritrocitos infectados; c) la determinación del volumen celular aglomerado mediante la técnica de microhematocrito (el cual puede ser de ayuda en la identificación de un proceso indicativo de anemia en caso de enfermedad); d) la realización de pruebas serológicas (que permitan identificar el tipo de respuesta inmune generada mediante la determinación de las proporciones de inmunoglobulinas igg1 e igg2, así como mediante la detección de citosinas características de la respuesta th1); e) la realización de pruebas moleculares (como la pcr anidada para el gen msp5) para la detección de adn del patógeno en sangre bovina.
De esta forma, el conjunto de estas evaluaciones permitiría determinar si la inoculación con péptidos sintéticos conteniendo los epítopos seleccionados cumple con el objetivo de estimular el desarrollo de una respuesta inmune protectora contra la anaplasmosis bovina causada por A. marginale en México.
Conclusión
Este tipo de trabajos in silico permite estimar la ubicación de los péptidos dentro de la conformación de las proteínas, con base en características como la hidrofobicidad o la antigenicidad, entre otras, de tal modo que se puede hacer una aproximación a la interacción que tendrían estas secuencias peptídicas con el sitio de reconocimiento de los anticuerpos. Utilizando herramientas bioinformáticas se logró predecir doce epítopos b, de los cuales se seleccionaron tres secuencias peptídicas que podrían ser consideradas para evaluarse como péptidos inmunogénicos contra la anaplasmosis bovina.
Referencias
Barigye, R., García Ortiz, M. A., Rojas Ramírez, E. E. y Rodríguez Camarillo, S. D. (2004). Identificación de antígenos igg2 específicos en tres cepas mexicanas de Anaplasma marginale. Técnica Pecuaria en Mexico, 42(2), 219-236. https://www.redalyc.org/pdf/613/61342208.pdf
National Library of Medicine (sf). Anaplasma marginale. National Library of Medicine. National Center of Biotechnology Information, sp. https://www.ncbi.nlm.nih.gov/datasets/genome/?taxon=770
Ramírez Rueda, R. Y. (2007). Búsqueda de secuencias con actividad inmunogénica, útiles para el diseño de un modelo de posible vacuna contra la malaria. Revista de la Universidad Industrial de Santander. Salud, 39(1), 8-15. https://www.redalyc.org/pdf/3438/343835674001.pdf
Rodríguez, S. D., García Ortiz, M. A., Jiménez Ocampo, R. y Vega y Murguía, C. A. (2009). Molecular epidemiology of bovine anaplasmosis with a particular focus in Mexico. Infection, Genetics and Evolution, 9(6), 1092-1101. https://doi.org/10.1016/j.meegid.2009.09.007
Rodríguez Camarillo, S. D., García Ortiz Ramón, M. A., Aboytes Torres, G. J. y Canto Alarcón, R. B. (2003). Inmunología e inmunoprofilaxis de la anaplasmosis bovina. Ciencia Veterinaria, 9(4), 123-164. https://www.fmvz.unam.mx/fmvz/cienciavet/revistas/CVvol9/CVv9c5.pdf
Salinas-Estrella, E., Amaro-Estrada, I., Cobaxin-Cárdenas, M. E., Preciado de la Torre, J. F. y Rodríguez, S. D. (2022). Bovine anaplasmosis: will there ever be an almighty effective vaccine? Frontiers in Veterinary Science, 9, 946545. https://doi.org/10.3389/fvets.2022.946545